
Genotipificación de aislados del complejo Mycobacterium tuberculosis provenientes del Hospital
Materno Infantil José Domingo de Obaldía, provincia de Chiriquí
Sara Miranda1,2
https://orcid.org/0000-0001-9235-1606
Fermín Acosta1
https://orcid.org/0000-0003-4849-9704
Amador Goodridge 1
https://orcid.org/0000-0003-3910-0482
1Instituto de Investigaciones Científicas y Servicios de Alta Tecnología de Panamá. Ciudad del Saber, Panamá
2Universidad Autónoma de Chiriquí, David, Chiriquí
Autor correspondiente: agoodridge@indicasat.org.pa
Enviado el: 9 de septiembre de 2023 Aceptado el: 10 de noviembre de 2023
https://doi.org/10.59722/rcvn.v1i2.703
Resumen
La tuberculosis (TB) es una enfermedad global infectocontagiosa de alta prioridad debido a su patogenicidad y facilidad de propagación. En Panamá, la TB, causada por Mycobacterium tuberculosis, presenta desafíos significativos, especialmente en provincias como Chiriquí, Colón, Panamá y las comarcas Guna Yala y Ngäbe-Buglé, donde la incidencia es elevada. El Hospital Materno Infantil José Domingo de Obaldía (HMIJDO) en Chiriquí juega un papel crucial en el diagnóstico y tratamiento de casos de TB, especialmente provenientes de la Comarca Ngäbe-Buglé y Bocas del Toro.
El HMIJDO facilita el aislamiento microbiológico de la cepa bacteriana M. tuberculosis, permitiendo la caracterización y genotipado de cepas implicadas en los casos de TB. La investigación se centra en caracterizar aislados del complejo M. tuberculosis del HMIJDO en 2017, 2019 y 2021, utilizando la reacción en cadena de la polimerasa con oligonucleótidos de alelo específico (ASO-PCR), un método molecular y genómico para rastrear cepas de alta transmisión.
Los resultados revelaron la presencia de cepas A del linaje 2 (3.5%), cepas B del linaje 4 genotipo Haarlem (2.4%), y cepas C del linaje 4 genotipo LAM (5.8%). Además, se identificaron cepas con resistencia a fármacos como rifampicina, isoniacida y MDR. Se detectaron posibles cepas mixtas, lo que complica el análisis y requiere pruebas adicionales.
El estudio proporciona una visión detallada de la distribución de los linajes de M. tuberculosis en la provincia de Chiriquí y áreas cercanas, identificando 10 cepas de M. tuberculosis, algunas asociadas con una mayor transmisión previamente registrada en Panamá y Colón. Este análisis contribuye a la comprensión de la epidemiología de la TB en la región y destaca la importancia de abordar la resistencia a fármacos y las cepas de alta transmisión para mejorar las estrategias de control de la enfermedad.
Palabras claves
ASO-PCR, cepas, cluster, CTAB, genotipificación, genotipos, linajes, Mycobacterium tuberculosis, tuberculosis.
Abstract
Tuberculosis (TB) is a high-priority global infectious disease due to its pathogenicity and ease of spread. In Panama, TB, caused by Mycobacterium tuberculosis, presents significant challenges, especially in provinces such as Chiriqui, Colon, Panama and the Guna Yala and Ngäbe-Bugle region, where the incidence is high. The Hospital Materno Infantil José Domingo de Obaldía (HMIJDO) in Chiriqui plays a crucial role in the diagnosis and treatment of TB cases, especially from the Ngäbe-Bugle and Bocas del Toro region.
The HMIJDO, facilitates the microbiological isolation of the bacterial strain M. tuberculosis, allowing the characterization and genotyping of strains implicated in TB cases. This research focuses on characterizing and isolating strains of M. tuberculosis complex gathered from HMIJDO in 2017, 2019, and 2021, using allele-specific oligonucleotide polymerase chain reaction (ASO-PCR), a molecular and genomic method for tracking high-transmission strains.
The results revealed the presence of strains A of lineage 2 (3.5 %), strains B of lineage 4 Haarlem genotype (2.4 %), and strains C of lineage 4 LAM genotype (5.8 %). In addition, strains with resistance to drugs such as rifampicin, isoniazid, and MDR were identified. Possible mixed strains were detected, which complicates the analysis and requires additional testing.
This study provides insights into the distribution of M. tuberculosis lineages in the province of Chiriqui and nearby areas, identifying 10 strains of M. tuberculosis, some of these associated with increased transmission previously reported in Panama and Colon. This analysis contributes to the understanding of TB epidemiology in the region and highlights the importance of addressing drug resistance and high-transmission strains to improve disease control strategies.
Keywords
ASO-PCR, cluster, CTAB, genotypes, genotyping, lineages, Mycobacterium tuberculosis, strains, tuberculosis.
Introducción
La tuberculosis (TB) es una enfermedad infecciosa originada por Mycobacterium tuberculosis agente etiológico de la TB en seres humanos, y en la actualidad es la principal causa de mortalidad a nivel global, cobrando aproximadamente 4000 vidas diarias (Organización Mundial de la Salud [OMS 2], 2020). En las últimas décadas, los estudios evolutivos han mejorado nuestra comprensión de la historia del complejo M. tuberculosis, enfatizando su larga coevolución con los seres humanos gracias a su habilidad para mantenerse en estado latente dentro del huésped.
Aunque el agente causal de la TB en seres humanos es la bacteria Mycobacterium tuberculosis (M. tuberculosis), integrante del Complejo M. tuberculosis; este complejo alberga diversas especies, diferenciadas por el huésped de infección, entre las que se incluyen Mycobacterium bovis (afectando ganados bovinos), Mycobacterium caprae (presente en cabras), Mycobacterium microti (relacionada con roedores), entre otras (Dorronsoro & Torroba, 2007).
Adicionalmente, hay otro grupo heterogéneo de bacterias que constituye, en numerosas ocasiones, la causa de infecciones en seres humanos, destacándose especies como Mycobacterium avium complex (MAC), Mycobacterium gordonae, Mycobacterium xenopi, Mycobacterium kansasii, Mycobacterium abscessus, Mycobacterium fortuitum y Mycobacterium chelonae, entre otras. Estas especies son conocidas como micobacterias no tuberculosas (MNT) o micobacterias atípicas que se aíslan con mayor frecuencia en humanos (Dorronsoro y Torroba, 2007; Porvaznik et al. 2017).
Sin embargo, el éxito patogénico principalmente de la especie M. tuberculosis se sustenta en su modalidad de infección y el desenvolvimiento de su patología en la forma pulmonar de la tuberculosis, ya que los pulmones constituyen su principal objetivo. Dicho proceso se compone de cuatro etapas secuenciales: la fagocitosis de bacilos, su reproducción intracelular, el estado latente de la infección y, finalmente, la activación de la infección pulmonar (Bañuls et al. 2015).
En Panamá, la TB exhibe elevadas tasas de incidencia (39.9 casos/100 000 hab.) y su epidemiología varía entre provincias y comarcas. La provincia de Chiriquí enfrenta desafíos y limitaciones en términos de diagnóstico, monitoreo de casos, tecnología y equipamiento para brindar atención adecuada a los pacientes de TB. La información concerniente a los genotipos de cepas circulantes en Chiriquí y regiones cercanas, como Bocas del Toro y la Comarca Ngäbe-Buglé, es limitada. Por lo tanto, se realizó un estudio de caracterización genotípica de cepas de M. tuberculosis obtenidas en Chiriquí, con el propósito de generar datos relevantes para el manejo de casos de TB y las cepas involucradas. La provincia de Chiriquí figura en la lista de regiones con elevada tasa de incidencia de TB, presentando un 25.3 % de la incidencia total de Panamá en una población de 100 000 habitantes. Es por ello que el objetivo central del estudio fue caracterizar las cepas aisladas de M. tuberculosis obtenidas del Hospital Materno Infantil José Domingo de Obaldía (HMIJDO) en Chiriquí durante los años 2017, 2019 y 2021 mediante técnicas moleculares y genómicas. Para ello, se emplearon técnicas de extracción de ADN utilizando la metodología con Bromuro de Cetiltrimetilamonio (CTAB) basada en solventes orgánicos y se caracterizaron las cepas de M. tuberculosis mediante la implementación de la reacción en cadena de la polimerasa con oligonucleótidos de alelos específicos (ASO-PCR).
Uno de los enfoques principales consistió en aplicar tres ASO-PCRs específicas (PCR A-Beijing, B-Haarlem y C-LAM) a muestras obtenidas de cultivos microbiológicos. Esta herramienta fue diseñada previamente para identificar cepas bacterianas de alta transmisión (A- Beijing, B- Haarlem y C- LAM) de M. tuberculosis en las provincias de Panamá y Colón. El propósito fue investigar la persistencia de la circulación de estas cepas a través del análisis retrospectivo de una colección de 85 cepas del HMIJDO en Chiriquí, las cuales fueron inactivadas utilizando métodos de bioseguridad de nivel 3 y posteriormente sometidas a extracción de ADN y genotipificación en el laboratorio del Centro de Biología Molecular y Celular del INDICASAT-AIP.
Materiales y Métodos
El estudio fue aprobado por el Comité de Bioética de la Universidad Santander de Panamá con los siguientes Protocolos: CBI-USantander-M-041-2022 y CBI-USantander-M-081-2022 (2022).
En el marco de nuestro estudio retrospectivo, las cepas del complejo M. tuberculosis, fueron obtenidas gracias la colaboración entre las instituciones INDICASAT-AIP y el HMIJDO para el estudio investigativo para entender la circulación de cepas de MTB en la región occidental. Nuestro estudio abordo el análisis de 85 cepas del Complejo M. tuberculosis recolectados y archivados durante un periodo de tres años: 2017, 2019 y 2021.
Obtención de cepas
De acuerdo al nivel de Bioseguridad 3, la colección de cepas de M. tuberculosis, forma parte del procedimiento estándar de cultivo y almacenamiento de aislados en el Laboratorio de Microbiología del Hospital Materno Infantil José Domingo de Obaldía, donde fueron desarchivadas e inactivadas en buffer de lisis, incubadas a 95°C por 20 minutos por el personal técnico del laboratorio del HMIJDO. Las muestras inactivadas fueron enviadas al INDICASAT-AIP para su posterior análisis en al área de Bioseguridad nivel 2. El número de aislados y las pruebas de sensibilidad fueron realizadas (en su momento) mediante método GenoType (Hain).
Genotipificación
La genotipificación completa de las cepas de M. tuberculosis se llevó a cabo en el Centro de biología celular y molecular de enfermedades (CBCME) del INDICASAT-AIP, que incluye los siguientes espacios
1-Extracción de ADN y cuantificación del material genético en nivel de Bioseguridad 2
· Se utilizó la técnica de extracción de ADN con Bromuro de Cetiltrimetilamonio (CTAB) que consiste en el
agregado de solventes orgánicos, procedimiento que consta de realizar toda la extracción en el mismo tubo de tapa rosca. Siguiendo la metodología elaborada por Doyle y Doyle, pero adaptada a este estudio, ya que en un principio se aplicó en la extracción de ADN para plantas, pero, ahora es ampliamente utilizado en la extracción de ADN de diversos microorganismos (Doyle y Doyle, 1987; Doyle, 1990).
· La cuantificación del ADN de M. tuberculosis se determinó evaluando la calidad y la cantidad del ADN utilizando el equipo NanoDrop™ 2000, Thermo Fisher Scientific, utilizando los estándares y manual del fabricante (Thermo fisher Scientific, 2010). La metodología consta de tomar 1 μl de la muestra de ADN y colocarla a en la celda de lectura para pedir la concentración en ng/ μl y la absorbancia 260/280.
2-PRE-PCR y POST-PCR= Implementación de las mezclas de reactivos para las tres ASO-PCRs (clústeres A, B y C)
· La caracterización genotípica de aislados de M. tuberculosis se realizó utilizando la metodología de la ASO-PCR, la cual se llevó a cabo siguiendo las condiciones descritas por Domínguez et al. (2019) presentadas en la tabla 1.
Tabla 1.
Mezclas de reacción y condiciones para cada PCR (A, B y C).
|
|
|
Volumen por reacción, µL |
|
|
|
Reactivo |
Cepa A |
Cepa B |
Cepa C |
|
|
HotStarTaq polimerasa, 5 U/µL diluido 1:10 |
1.25 |
1.25 |
1.25 |
|
|
MgCl2, 25 mM |
0.4 |
0.3 |
0.4 |
|
|
Búffer de PCR, 10µcon 15 mM MgCl2 |
2.5 |
2.5 |
2.5 |
|
|
Solución Q, 5µ |
5.0 |
5.0 |
5.0 |
|
|
Mezcla de cebadores, 10µ |
2.5 |
2.5 |
2.5 |
|
|
dNTP, 10 mM |
0.2 |
0.2 |
0.2 |
|
|
H2O Diana de ADN |
10.65 |
10.75 2.5 µL c/u |
10.7 |
|
|
Volumen de reacción final de 25 µL |
|
|
|
|
Nota: *dNTPs, desoxinucleótidos; MgCl2, Cloruro de magnesio de magnesio. Las condiciones de PCR para cada cepa fueron las siguientes: 95° C durante 15 min, seguido de 27 ciclos de 95°C durante 1 min, recocido (64°C para Cluster A y 60 °C tanto para Cluster B como C durante 1 min) y 72°C durante 10 min.
La Post-PCR consiste del procedimiento de la electroforesis, donde los amplicones obtenidos se analizaron mediante electroforesis en gel de agarosa al 3 % en 100 ml de Buffer TBE, con tinción de bromuro de etidio (6 μl). Una vez el gel solidificado se carga la muestra de ADN con un volumen de 12 μl en cada uno de los pocillos del gel de agarosa; estas se mezclaron con 4 μl de Buffer o tinte de carga: Blue/Orange Loading Dye, 6X de Promega. Los tamaños de los amplicones se estimaron por comparación con marcadores de peso molecular de la escalera de ADN de 100 pb, PROMEGA. Posteriormente, las electroforesis, corrieron a 95V durante una hora y media y fueron visualizadas en un Transiluminador ultravioleta de CHEMIDOC.
Finalmente, el análisis de fragmentos se realizó después de la electroforesis, constaba de tomar el gel de agarosa y colocarlo en el Transiluminador (CHEMIDOC), en la bandeja lectora del gel #2 que tiene la especificidad para revelar gel de garosa con tinte de Bromuro de etidio. Una vez realizada la lectura se muestra en la pantalla el gel con las bandas reflejadas. El análisis se lleva a cabo comparando el tamaño de la banda con el control positivo de cada Cluster (Cluster A, Cluster B y Cluster C) además del control negativo la cepa H37RV y así determinar el tipo de cepa. El tamaño de la banda se corroboraba con la escalera de 100 bp (Promega).
Resultados
En el transcurso de este estudio, se logró acceder a una colección compuesta por un total de 85 cepas de M. tuberculosis. Estas cepas se distribuyen en tres años distintos de recolección: 22 cepas provienen del año 2017, 55 cepas del 2019 y finalmente, 8 cepas del año 2021; a todas estas cepas, se llevó a cabo un análisis microbiológico de su susceptibilidad a fármacos. Se determinó la presencia de drogorresistencia en las 85 cepas de M. tuberculosis estudiadas, dando como resultado la identificación de dos cepas correspondientes a casos de monoresistencia; distribuidas de la siguiente manera:
· 1 cepa mostró resistencia únicamente a Rifampicina.
· 1 cepa demostró resistencia exclusiva a Isoniacida.
Resulta relevante destacar que estas dos cepas monorresistentes fueron aisladas y obtenidas de la Provincia de Chiriquí. No se encontraron cepas monorresistentes entre los 12 aislados provenientes de la Comarca Ngäbe-Buglé ni los nueve aislados de Bocas del Toro. Por lo tanto, se puede concluir que estos números específicos a las regiones corresponden a cepas sensibles a los fármacos antituberculosos.
Por otro lado, en relación a la identificación de cepas multidrogorresistentes (MDR), se obtuvo el resultado de que entre los 85 aislados totales, solamente tres cepas corresponden a casos de MDR, es decir, cepas resistentes tanto a Rifampicina como a Isoniacida. Estas cepas MDR están distribuidas entre los 58 aislados de la provincia de Chiriquí. En cambio, no se lograron identificar cepas MDR en las muestras provenientes de la Comarca Ngäbe-Buglé ni de Bocas del Toro.
En última instancia, dentro de este conjunto de 85 aislados, se asignó una categoría particular denominada "Datos no disponibles". En esta categoría se incluyen seis aislamientos sometidos a pruebas de resistencia a fármacos. Sin embargo, debido a problemas técnicos en el formulario de inscripción, no se reflejó adecuadamente la provincia a la que pertenecían los pacientes en cuestión.
Tabla 2.
Resistencia de los aislados M. tuberculosis obtenidas en el Hospital materno infantil José domingo de Obaldía, Chiriquí (N°85).
|
Región |
N° de aislados |
Monoresistente |
MDR |
Sensibilidad |
No data |
|
Chiriquí |
58 |
2 (RR, IR) |
3 |
53 |
0 |
|
Comarca Ngäbe-Buglé |
12 |
0 |
0 |
12 |
0 |
|
Bocas del Toro |
9 |
0 |
0 |
9 |
0 |
|
Datos no disponibles |
6 |
0 |
0 |
5 |
1 |
Nota: R-R (resistente Rifampicina), R-I (resistente Isoniacida), MDR (Multidrogoresistente)
* Datos no disponible: Datos no llenados en el formulario del paciente, por ejemplo, la región donde reside.
Hallazgos de las Tres ASO-PCR
En esta sección, se exponen los resultados obtenidos a través de la implementación de las tres ASO-PCR (ASO-PCR A-Beijing, B-Haarlem y C-LAM). La colección compuesta por 85 aislados fue sometida a los tres ensayos de PCR con el propósito de identificar y caracterizar los genotipos más predominantes pertenecientes a los linajes 2 y 4, los cuales habían sido previamente señalados como problemáticos en las áreas de Panamá y Colón. De los 85 aislamientos recolectados durante los tres años mencionados, fue posible llevar a cabo la caracterización genotípica de manera exitosa en 10 de estas cepas, las cuales fueron clasificadas como parte de los Cluster específicos:
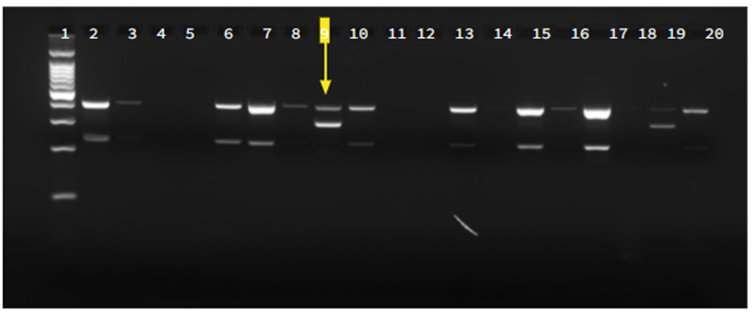
Figura 1.
Resultados de cepa A (Linaje 2, genotipo Beijing). Electroforesis en gel de agarosa al 2% para la identificación de la cepa de Cluster A (linaje2 genotipo Beijing). El pocillo 1 representa la escalera/marcador de peso molecular de 100 pb PROMEGA, el pocillo 2 al 18 representan las muestras de ADN amplificadas, la flecha del pocillo 9 indica la presencia de la cepa A, el pocillo 19 y 20 representan los controles utilizados (Pocillo 19: control positivo y pocillo 20 el control negativo).
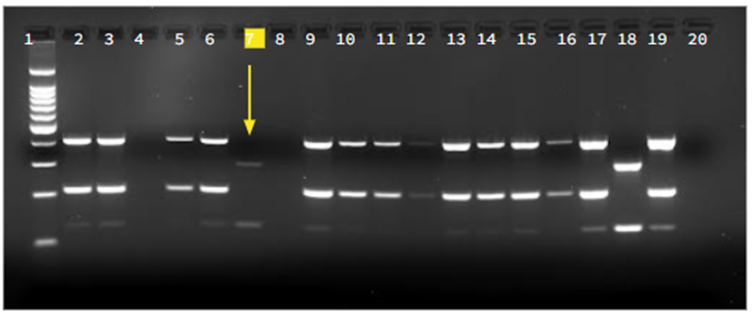
Figura 2.
Resultados de cepa B (Linaje 4 genotipo Haarlem). Electroforesis en gel de agarosa al 2% para la identificación de la cepa de Cluster B (linaje 4 genotipo Haarlem). El pocillo 1 representa la escalera de
peso molecular de 100 pb PROMEGA, el pocillo 2 al 17 representan las muestras de ADN amplificadas, la flecha del pocillo 7 indica la presencia de la cepa B, el pocillo 18 y 19 representan los controles utilizados (Pocillo 18: control positivo y pocillo 19 el control negativo). Por último, el pocillo 20 se le agregó agua como control de contaminación.
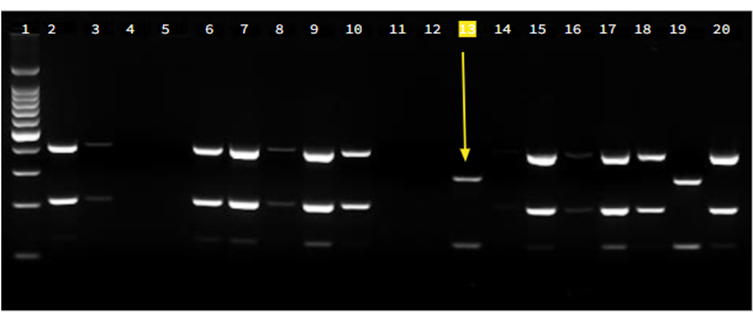
Figura 3.
Resultados de cepa B (Linaje 4 genotipo Haarlem). Electroforesis en gel de agarosa al 2% para la identificación de la cepa de Cluster B (linaje 4 genotipo Haarlem). El pocillo 1 representa la escalera de peso molecular de 100 pb PROMEGA, el pocillo 2 al 18 representan las muestras de ADN amplificadas, la flecha del pocillo 13 indica la presencia de la cepa B, el pocillo 19 y 20 representan los controles utilizados (Pocillo 19: control positivo y pocillo 20 el control negativo).
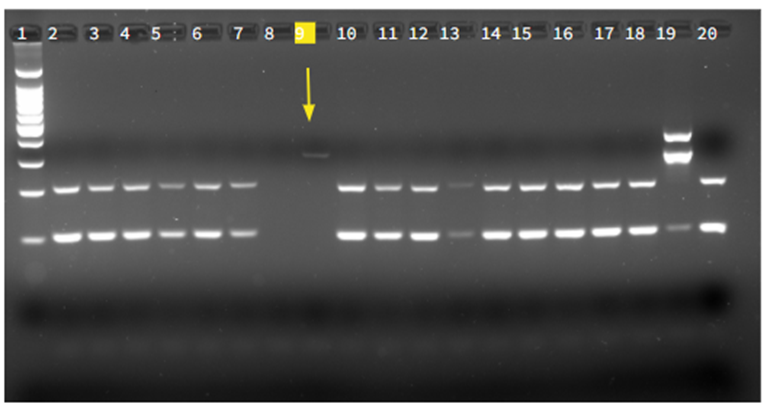
Figura 4.
Resultados de cepa C (Linaje 4 genotipo LAM). Electroforesis en gel de agarosa al 2% para la identificación de la cepa de Cluster C (linaje 4 genotipo LAM). El pocillo 1 representa la escalera de peso molecular de 100 pb PROMEGA, el pocillo 2 al 18 representan las muestras de ADN amplificadas, la flecha del pocillo 9 indica la presencia de la cepa B, el pocillo 19 y 20 representan los controles utilizados (Pocillo 19: control positivo y pocillo 20 el control negativo).

Figura 5.
Resultados de cepa C (Linaje 4 genotipo LAM). Electroforesis en gel de agarosa al 2% para la identificación de la cepa de Cluster C (linaje 4 genotipo LAM). El pocillo 1 representa la escalera de peso molecular de 100 pb PROMEGA, el pocillo 2 al 18 representan las muestras de ADN amplificadas, la flecha del pocillo 9 indica la presencia de la cepa B, el pocillo 19 y 20 representan los controles utilizados (Pocillo 19: control positivo y pocillo 20 el control negativo).
Tabla 3.
Resumen de resultados de las tres ASO-PCRs aplicadas sobre la colección de 85 aislados de del complejo M. tuberculosis.
|
ASO-PCR |
CEPA (%) |
NO CEPA (%) |
|
CEPA-A (Beijing) |
3 (3.5) * |
82 (96.4) |
|
CEPA-B (Haarlem) |
2 (2.3) |
83 (97.6) |
|
CEPA-C (LAM) |
5 (5.8) ** |
80 (94.1) |
LAM: Latin America Mediterraneo, *Dentro del total de estas tres CEPAS A identificadas, dos muestras son mixtas. ** Del total de las CEPA C, dos muestras son mixtas.
Discusión
La transmisión de la TB es un tema global que requiere ser atendido en todo su aspecto. El abordaje de los distintos genotipos implicados en los casos de TB utilizando las herramientas de epidemiología molecular ha resultado útil para tales escenarios, permitiendo conocer la transmisión de las cepas más prevalentes o activas (Coll y García de Viedma, 2018).
En este sentido, la aplicación de herramientas de vigilancia epidemiológica como la PCR alelo específicas (ASO-PCR) ha simplificado los pasos en la identificación de cepas de alta transmisión o de mayor riesgo en Panamá y en otras regiones.
Teniendo en cuenta esto, se utilizaron herramientas de microbiología, biología molecular y genética en la provincia de Chiriquí con la necesidad de conocer la transmisión de cepas problemáticas o prevalentes del complejo M. tuberculosis dirigimos nuestro enfoque en el abordaje de análisis retrospectivo de una colección cepas colectadas en el HMIJDO, provincia de Chiriquí. La colección de 85 cepas del HMIJDO representa aproximadamente el 10% del total de los casos entre las tres regiones nivel del total de los casos de TB que recibe el hospital HMIJDO entre las tres regiones (Bocas del Toro, Comarca Ngäbe-Buglé y Chiriquí). De todos los casos reportados de TB durante los años 2017, 2019 y 2021, tuvimos acceso a 85 aislados repartidos entre las tres regiones como la provincia de Chiriquí Bocas del toro y la Comarca Ngäbe Buglé.
Análisis de la calidad de ADN de bacterias
En esta primera etapa nuestro punto de partida fue obtener la mayor cantidad de ADN posible utilizando el método de extracción de CTAB basado en solventes orgánicos, debido a que ofrece excelentes resultados y bajo costo (Huang, et al. 2018). Tiene la capacidad de analizar Micobacterias que se caracterizan por su resistencia a nivel de membrana, proteínas y lípidos, (Wilson, 2001). Nuestro análisis cuantitativo de ADN arrojó un promedio aproximado de 30 ng/ml y una absorbancia de 1.60 nm, valores aceptables durante la medición de acuerdo a los valores óptimos indicadas en el manual del equipo (Thermo Scientific NanoDrop Spectrophotometers, 2010). Este método de extracción por CTAB demostró tener buen rendimiento durante el análisis de ADN. Es notorio mencionar que la cantidad de ADN a extraer dependerá de la cantidad del material inicial de trabajo, por lo que es necesario almacenar una cantidad de material necesario de cultivos que pudieran ser de potencial estudio en posteriores estudios como es el caso de subcultivo, extracción de DNA, análisis de genoma completo y otros ensayos que pudieran ser de interés.
Genotipificación de cepas de MTB prevalentes mediante PCR alelo específico (ASO-PCR)
Es una técnica que pretende simplificar la vigilancia epidemiológica de los casos de TB. En este caso, dirigimos esfuerzos en su aplicación a los aislados de M. tuberculosis obtenidas en la región occidental
de Panamá con la finalidad de abordar y proveer nuevos datos para mejorar la compresión de la transmisión de la TB mediante el uso del método evaluado y diseñado previamente en Panamá y Colón (Domínguez et al. 2019). Con el objetivo de identificar la presencia de cepas de M. tuberculosis prevalentes y activa en la provincia de Chiriquí, aplicamos tres PCR específicas para identificar Cluster A (cepa linaje 2, genotipo Beijing prevalente), Cepa B (linaje 4, genotipo Haarlem prevalente) y Cepa C (linaje 4, genotipo LAM de transmisión activa).
La primera ASO-PCR aplicada tiene la capacidad de caracterizar genotípicamente cepas de M. tuberculosis del Linaje 2 genotipo Beijing. Este linaje geográficamente se distribuye por toda Asia, pero característicamente el genotipo bacteriano Beijing es considerado actualmente uno de los linajes más prevalentes de M. tuberculosis siendo reportado en diversos países en todo el mundo (Mokrousov, 2016). Es de interés de estudio debido a que su prevalencia se debe a su alta transmisibilidad y virulencia hasta el punto de dispersarse por toda Latino América con gran adaptación a dicha población (Mokrousov, 2016). En nuestro estudio pudimos destacar la presencia de tres cepas correspondiente al genotipo Beijing (figura 1), que representó el 3.5 % del total de las muestras analizadas (n: 85). Aunque esta cifra es baja en comparación con la provincia de Colón, donde el genotipo Beijing llega a representar hasta el 44.1 % de los casos de tuberculosis (Acosta et. al 2020). Esto sigue siendo una señal importante para evaluar la epidemiología de la tuberculosis en Chiriquí y sus regiones aledañas, resulta crucial investigar a estos pacientes de genotipo Beijing y conocer su contacto con la población de Panamá y Colón y, evaluar si es un caso de transmisión independiente como ha ocurrido en otros entornos (López-Rocha et al. 2013).
Por otro lado, la aplicación de la segunda ASO-PCR tiene la capacidad de caracterizar genotípicamente el linaje 4 identificando las cepas B de transmisión prevalente y cepa C de transmisión activa del linaje 4 de M. tuberculosis. Nuestro estudio permitió identificar dos cepas B (genotipo Haarlem), representado el 2.3 % (figuras 2 y 3), mientras que la cepa C (genotipo LAM) fue del el 5.8 % del total de 85 muestras analizadas (figuras 4 y 5). Estos resultados indican que los genotipos de las cepas B y C se transmiten con baja frecuencia en la zona occidental (tabla 3), lo que sugiere la existencia de otros genotipos involucrados en los casos de tuberculosis. De acuerdo a Demay et al. (2012) confirma que los genotipos Haarlem y LAM del Linaje 4 son los miembros más comúnmente observados en América del Sur, América Central y el Caribe aparte de estar distribuidos en Europa y África Central. Teniendo en cuenta esto es lógico haber encontrado en mayor proporción el genotipo Haarlem y LAM en este estudio, por lo que se puede tener certeza de que el linaje 4 está distribuido en toda América y nivel mundial; y que todos los aislados analizados en diversos estudios incluyendo este se van a detectar los genotipos Haarlem y LAM.
En cuanto al resto de las muestras micobacterianas que no se clasificaron en Cepa A, Cepa B y Cepa C, la razón es atribuida a que los amplicones visualizados en el gel eran distintos al control positivo del Cluster A para identificar cepas de dicho Cluster del L2-genotipo Beijing, y así mismo, los amplicones eran diferentes a cepas del Cluster B para identificar L4-genotipo Haarlem y cepas del Cluster C para L4-genotipo LAM por lo que no se pudieron identificar el genotipo de M. tuberculosis, por lo tanto, tampoco se conoce el linaje, sin embargo existe la seguridad que son cepas de la especie de M. tuberculosis ya que sus amplificaciones eran iguales a la cepa micobacteriana modelo H37Rv. Esto señala que no se pudo determinar la identidad genotípica de estas cepas debido a que el análisis se enfocó en las tres cepas de mayor prevalencia descritas en estudios previos en Colón y Panamá, que son responsables de los casos de tuberculosis (Domínguez et al. 2019).
Entorno epidemiológico
Los resultados obtenidos revelan que la epidemiología de la provincia de Chiriquí difiere de la de otras provincias de Panamá. Estos hallazgos sugieren que las cantidades de genotipos reportados no son iguales para considerar que la epidemiología es homogénea, como lo es para el genotipo Beijing en las provincias de Panamá y Colón. Estos datos indican que la prevalencia de la tuberculosis en la región occidental se debe a las condiciones socioambientales y a la calidad de vida de las personas. En el caso de Chiriquí, la presencia de zonas desatendidas es notable, debido a la dificultad de acceso a estas regiones y a la falta de suministros básicos, lo que conduce a problemas como la desnutrición y la limitación del acceso a agua potable, afectando la higiene personal y ambiental. Esto podría aumentar la prevalencia de otras micobacterias (López et al. 2021). Las áreas más afectadas por estas circunstancias son la Comarca y las regiones circundantes. La Comarca Ngäbe Buglé, con una población de 213,860 habitantes, no cuenta con la atención médica necesaria debido a la falta de instalaciones de salud adecuadas y equipamiento para el diagnóstico de la tuberculosis. Solo unos pocos habitantes tienen la posibilidad de viajar al HMIJDO para recibir atención médica y diagnóstico confiable.
En el estudio, de las 85 muestras analizadas, solo 12 corresponden a la Comarca (tabla 2), lo que no significa que haya pocos casos de tuberculosis en esa población, si comparamos esta cifra con los datos actualizados del HMIJDO, queda claro que la tuberculosis es la principal causa de ingreso hospitalario en todas las edades, especialmente en la población infantil. La falta de medicamentos específicos para cada paciente es otro problema importante, ya que el tratamiento inadecuado o la ausencia de medicación desarrollan el riesgo de cepas resistentes a los fármacos, aumentando la vulnerabilidad e inmunidad del huésped y el riesgo social de la población (Afshar et al. 2019, Gadoev et al. 2017).
En este estudio, identificamos cinco cepas con resistencia a los fármacos, que se dividen en dos cepas monorresistentes a rifampicina e isoniazida, y tres cepas MDR. La falta de medicamentos adecuados y la discontinuidad en el programa antituberculoso contribuyen a esta situación, ya que lamentablemente no se rigen a la norma técnica nacional para el control de la tuberculosis en Panamá (Ministerio de Salud [MINSA], 2017). La presencia de estas cepas resistentes es un desafío considerable en los esfuerzos por erradicar la tuberculosis, y subraya la importancia de identificar las cepas de M. tuberculosis MDR (Organización Mundial de la Salud [OMS 2], 2020).
Por otro lado, durante el transcurso de esta investigación surgieron ciertas limitaciones, como la disponibilidad limitada de cepas proporcionadas por el HMIJDO debido a la pandemia de COVID-19 (OMS 1, 2020). Afectando el flujo de trabajo en microbiología y la logística de envío de cepas. Además, notamos un bajo número de muestras, lo que restringió la posibilidad de repetir tanto los análisis moleculares como los genómicos. En la etapa de análisis de la ASO-PCR, también detectamos cepas mixtas, que son infecciones con más de un genotipo, lo que puede complicar los resultados del tratamiento. Sin embargo, debido a la falta de acceso al medio de cultivo bacteriano, no pudimos resolver este problema (Kargarpour Kamakoli et al. 2020).
En última instancia, este estudio proporciona información valiosa sobre la epidemiología de la tuberculosis en la provincia de Chiriquí, que hasta ahora no había sido estudiada en profundidad en comparación con otras provincias de Panamá. Esta información es esencial para la provincia, el país y a nivel internacional, ya que contribuye al conocimiento actualizado sobre la bacteria y sus características microbiológicas, así como a las medidas necesarias para su diagnóstico.
Conclusiones
· Este estudio permitió evidenciar la distribución de los linajes de aislamientos del complejo bacteriano de M. tuberculosis circulantes en la región occidental de Panamá. Se lograron identificar 10 cepas de M. tuberculosis distribuidas en la población de la provincia de Chiriquí y regiones cercanas de Bocas del Toro y Comarca Ngäbe Buglé; cepas encontradas con genotipos de mayor transmisión descritos previamente en Panamá y Colón.
· Con la información obtenida de los resultados de esta investigación se puede evidenciar que el Linaje 4 de M. tuberculosis es el de mayor transmisibilidad en la provincia de Chiriquí, donde se destaca la presencia del genotipo LAM.
· Hay una escasa predominancia de variantes genéticas problemáticas (cepa con genotipo Beijing) responsables de la enfermedad ya que no encontramos en la colección analizada de la región occidental la misma proporción de cepas problemáticas que en el resto del país (provincia de Panamá y Colón), posiblemente por la diferencia de entornos, diversas condiciones de vida, zona ambiental y cultural que lo hace limitante a que esa cepa se disperse.
· Los descubrimientos indican la necesidad de llevar a cabo investigaciones prospectivas con un enfoque sistémico, que relacionen los casos de tuberculosis con determinantes socioeconómicos y demográficos, con el propósito de lograr una comprensión más exhaustiva de su comportamiento.
· Es esencial emprender análisis de secuenciación completa de genomas de las cepas, con el fin de establecer vínculos genómicos a lo largo del tiempo y detectar nuevas instancias de resistencia a antimicrobianos.
· Definitivamente, este estudio adquiere relevancia primordial en el panorama investigativo y académico, puesto que contribuye de manera sustantiva a la identificación de cepas bacterianas con implicancias problemáticas y profundiza en la comprensión de su dinámica de transmisión y resistencia.
Agradecimientos
A todas las entidades y organizaciones involucradas: UNACHI, INDICASAT-AIP, Hospital materno infantil José Domingo de Obaldía, MINSA, Apoyo por Fondos de Investigación: Sistema Nacional de Investigación
(SNI) de la SENACYT, Fondos internos del INDICASAT, Red de Países de la Unión Europea (UE), América Latina y el Caribe (ALC)-ERANET-LAC y Beca mediante la Fundación Deveaux.
Referencias
Acosta, F., Norman, A., Sambrano, D., Batista, V., Mokrousov, I., Shitikov, E., Jurado, J., Mayrena, M., Luque, O., Garay, M., Solís, L., Muñoz, P., Folkvardsen, D. B., Lillebaek, T., Pérez-Lago, L., Goodridge, A., García de Viedma, D. (2020). Prevalencia probable a largo plazo de un clon predominante de Mycobacterium tuberculosis de un genotipo Beijing en Colón, Panamá.
Afshar, B., Carless, J., Roche, A., Balasegaram, S., & Anderson, C. (2019). Surveillance of tuberculosis (TB) cases attributable to relapse or reinfection in London, 2002-2015. PloS one, 14(2), e0211972. https://doi.org/10.1371/journal.pone.0211972
Bañuls, A. L., Sanou, A., Van Anh, N. T., & Godreuil, S. (2015). Mycobacterium tuberculosis: ecology and evolution of a human bacterium. Journal of medical microbiology, 64(11), 1261–1269. https://doi.org/10.1099/jmm.0.000171
Coll, D. García de Viedma. (2018). Epidemiología molecular de la tuberculosis. Elsevier España, S.L.U. y Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica.
Demay, C., Liens, B., Burguière, T., Hill, V., Couvin, D., Millet, J., Mokrousov, I., Sola, C., Zozio, T., Rastogi N. (2012). SITVITWEB--a publicly available international multimarker database for studying Mycobacterium tuberculosis genetic diversity and molecular epidemiology. Infect Genet Evol. 12(4):755–66. https://doi.org/10.1016/j.meegid.2012.02.004
Doyle, J., & Doyle, J. (1987). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical bulletin, 19:11–15.
Doyle, J. (1990). DNA protocols for plants. Nato Asy Series, 283-284.
Domínguez, J., Acosta, F., Pérez-Lago, L., Sambrano, D., Batista, V., De La Guardia, C., Abascal, E., Chiner-Oms, Á., Comas, I., González, P., Bravo, J., Del Cid, P., Rosas, S., Muñoz, P., Goodridge, A., & García de Viedma, D. (2019). Simplified Model to Survey Tuberculosis Transmission in Countries Without Systematic Molecular Epidemiology Programs. Emerging infectious diseases, 25(3), 507–514. https://doi.org/10.3201/eid2503.181593
Dorronsoro, I., & Torroba, L. (2007). Microbiología de la tuberculosis. Anales del Sistema Sanitario de Navarra, 30(Supl. 2), 67-85. Recuperado en 10 de noviembre de 2023, de http://scielo.isciii.es/scielo.php?script=sci_arttext&pid=S113766272007000400006&lng=es&tlng=es.
Gadoev, J., Asadov, D., Harries, A. D., Parpieva, N., Tayler-Smith, K., Isaakidis, P., Ali, E., Hinderaker, S. G., Ogtay, G., Ramsay, A., Jalolov, A., & Dara, M. (2017). Recurrent tuberculosis and associated factors: A five-year countrywide study in Uzbekistan. PloS one, 12(5), e0176473. https://doi.org/10.1371/journal.pone.0176473
Huang, X., Duan, N., Xu, H., Xie, T. N., Xue, Y. R., & Liu, C. H. (2018). Molekuliarnaia biologiia, 52(4), 718–726. https://doi.org/10.1134/S0026898418040080
Kargarpour Kamakoli, M., Farmanfarmaei, G., Masoumi, M., Khanipour, S., Gharibzadeh, S., Sola, C., Fateh, A., Siadat, S. D., Refregier, G., & Vaziri, F. (2020). Prediction of the hidden genotype of mixed infection strains in Iranian tuberculosis patients. International Journal of infectious diseases: IJID: official publication of the International. Society for Infectious Diseases, 95, 22–27. https://doi.org/10.1016/j.ijid.2020.03.056
López, A., Acosta, F., Sambrano, D., Tarajia, M., Navajas, S., Arias, F., Escobar, B., Ortis, P., Adames, F., & Goodridge, A. (2021). Caracterización Molecular Directa del Frotis de Bacilos Ácido-Resistentes de Especies de Mycobacterium NoTuberculosis 96 Causantes de Tuberculosis Pulmonar en la Región Guna Yala, Panamá. The American Journal of tropical medicine and hygiene, 105(3), 633–637. https://doi.org/10.4269/ajtmh.21-0096
López-Rocha, E., Juárez-Álvarez, J., Riego-Ruiz, L., Enciso-Moreno, L., Ortega-Aguilar, F., Hernández-Nieto, J., Enciso-Moreno, J. A., & López-Revilla, R. (2013). Genetic diversity of the Mycobacterium tuberculosis complex in San Luis Potosí, México. BMC research notes, 6, 172. https://doi.org/10.1186/1756-0500-6-172
Ministerio de Salud. [MINSA]. (2017). Norma técnica nacional para el control de la tuberculosis. Revisión y actualización de las Normas y Procedimientos para la Prevención y Control de la Tuberculosis. https://www.minsa.gob.pa/sites/default/files/programas/10sept_norma_tb_digital.pdf
Mokrousov I, Vyazovaya A, Iwamoto T. (2016). Linaje latinoamericano-mediterráneo de Mycobacterium tuberculosis: rastros humanos a través de la filogeografía de patógenos. Mol Phylonet Evol . 99 :133–143.
Organización Mundial de la Salud. [OMS 1]. (2020). La OMS caracteriza a COVID-19 como una pandemia. https://www.paho.org/es/noticias/11-3-2020-oms-caracteriza-covid-19-como-pandemia
Organización Mundial de la Salud [OMS 2]. (2020) Directrices consolidadas de la OMS sobre tuberculosis Módulo 4: Tratamiento de la tuberculosis resistente a los medicamentos. Ginebra.
Porvaznik, I., Solovič, I., & Mokrý, J. (2017). Non-Tuberculous Mycobacteria: Classification, Diagnostics, and Therapy. Advances in experimental medicine and biology, 944, 19–25. https://doi.org/10.1007/5584_2016_45
Thermo Scientific NanoDrop Spectrophotometers. (2010). Nucleic Acid. https://www.thermofisher.com/documentconnect/documentconnect.html?url=https%3A%2F%2Fassets.thermofisher.com
%2FTFS- Assets%2FCAD%2Fmanuals%2Fts-nanodrop-nucleicacid-olvr2.pdf
Wilson K. (2001). Preparation of genomic DNA from bacteria. Current protocols in molecular biology, Chapter 2. https://doi.org/10.1002/0471142727.mb0204s56